特長 > 距離を測定する
距離を測定する
コンフォメーション変化で動いた距離を測る
状態の異なる2つの構造のコンフォメーション変化を見る時には、それらの構造を重ね合わせます。重ね合わせた状態で距離を測定すれば、着目する残基がどのくらい変化したのか調べることができます。
距離表示を編集する
距離を表示した後で、立体構造を動かすと、距離表示も一緒に移動します。選択して、好みの位置に移動する事もできます。点線のみの表示にしたり、文字のフォントや色、大きさを変えられますので、作図の際にも便利です。
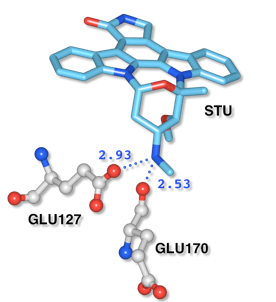
Protein kinase Aと阻害剤スタロスポリンの複合体(1STC)のスタウロスポリンの窒素原子(N4)からの距離測定の例
Chain Interfaceでは、蛋白質やDNAのチェイン間で相互作用している残基を網羅的に検出することができます。
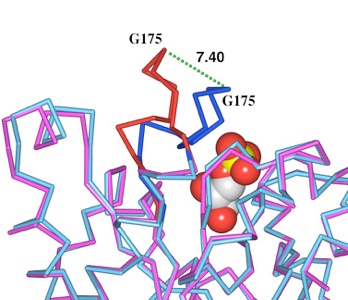
コンフォメーション変化を見るには、Auto Fitによる自動重ね合わせが便利です。
Triose-phosphate Isomerase (6TIM) のフリーの状態 (Open)
と阻害剤が結合した状態 (Close) をAuto-fitで重ね合わせた
もの。Gly175のCαが7.40Åも動いていることがわかります。
近いか遠いか、数値で確認。
結果や考察では、原子間距離を数値で示すことが重要な場合があります。Waalsなら立体構造を見ながら二点の原子をクリックするだけで距離を測定できます。
図に示す事で、表示だけでは伝わらない原子間距離が明確に。距離表示は、色や大きさを変えたり、表示する位置を移動したりすることもできるので、そのまま図として使えます。
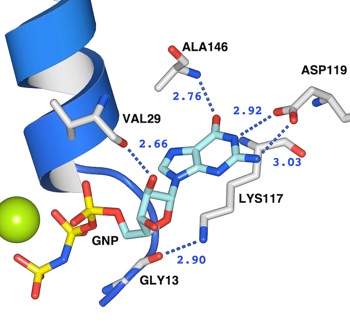
阻害剤とアミノ酸残基の距離を測定する
タンパク質に結合している阻害剤や基質アナログと、アミノ酸残基の距離を測定して結合の様子を確認できます。
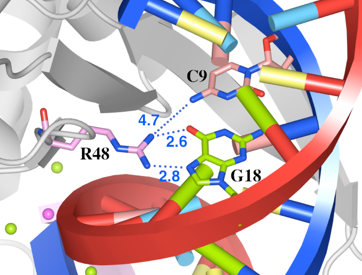
チェイン間のアミノ酸残基どうしの距離を測定する
タンパク質のチェイン間での2つのアミノ酸残基や、DNAとの複合体でのアミノ酸残基と塩基などが相互作用しているかどうか、距離を測定して確認できます。
Waalsの特長

タンパク質分子のためのインタフェース
見る・調べる
もっと便利に
保存する
動画を作成する
作図する
分子間の距離関係を調べる
重ね合わせと比較

蛋白質のためのインタフェース
Waalsの特長
見る・調べる
作図する
分子間の距離関係を調べる
重ね合わせと比較
もっと便利に
保存する
動画を作成する
変異体の解析に
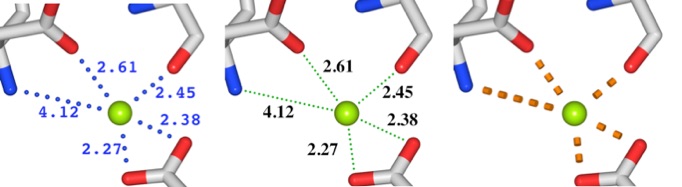
変異によるアミノ酸置換の影響を調べるとき、アミノ酸置換によってリガンドとの結合がどう変わったか、距離を測定して調べることができます。
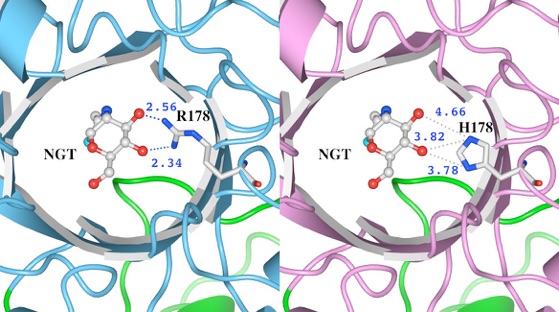
Hexosaminidase Aのαで起こった変異の一つを調べた例です。野生型R178では、窒素原子とリガンドの間に水素結合可能な距離が二カ所ありますが、変異体のH178ではリガンドとの距離がいずれも水素結合可能な範囲にないことがわかります。

距離測定ツール
Copyright © 2009-2025 Altif Laboratories Inc. All rights reserved.