関連情報 SWISS-MODEL の使い方 > Automated Modeでホモロジーモデリングを行う
SWISS-MODEL Automated Modeでホモロジーモデリングを行う 1
以下の入力画面が開かれます。各項目に入力します。ここでは、Dual specificity mitogen-activated protein kinase kinase 3(UniProt Accession Code:P46734, MP2K3_HUMAN) の構造モデルを作成します。
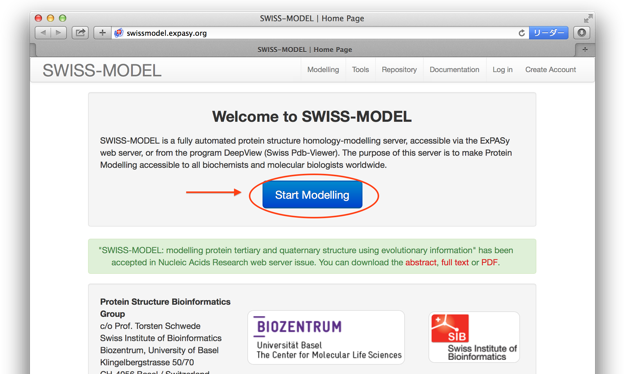
SWISS-MODELでは、Webサイト上でホモロジーモデリング法により、アミノ酸配列の相同性が高いタンパク質の立体構造をテンプレートにして立体構造モデルを構築することができます。リガンドが結合している構造をテンプレートに指定することで、SWISS-MODELの基準を満たしていれば、リガンドが結合した状態の構造モデルを構築することも可能です。ここでは、「Automated Mode」でのモデリングの手順についてご紹介します。SWISS-MODELはSwiss Institute of Bioinformaticsにより提供されています。ご利用にあたっては、SWISS-MODELのサイトより内容をご確認の上お使い下さいますようお願い申し上げます。SWISS-MODELの参考文献については、 https://swissmodel.expasy.org/docs/references/ をご参照下さい。
1. SWISS-MODEL のトップページを開く
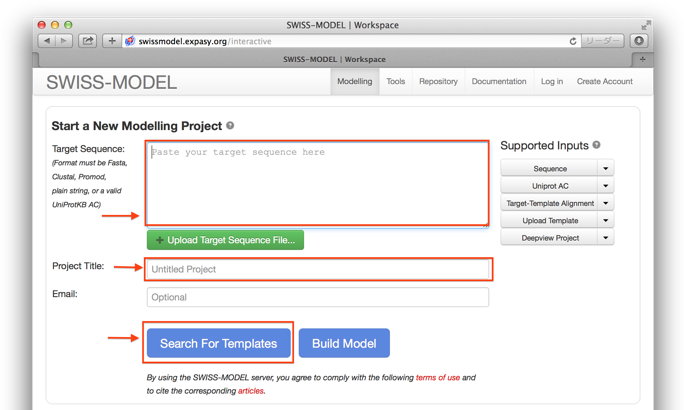
2. 入力画面で対象タンパク質のアミノ酸配列を入力し、テンプレート構造を検索する。
-
(1)「Target Sequence」に作製したいモデルのアミノ酸配列(Fasta形式)あるいは、UniProtのAccession Codeを入力します。ここでは、ACのP46734を入力します。手元のアミノ酸配列のファイルをアップロードする場合は、「Upload Target Sequence File..」をクリックします。
-
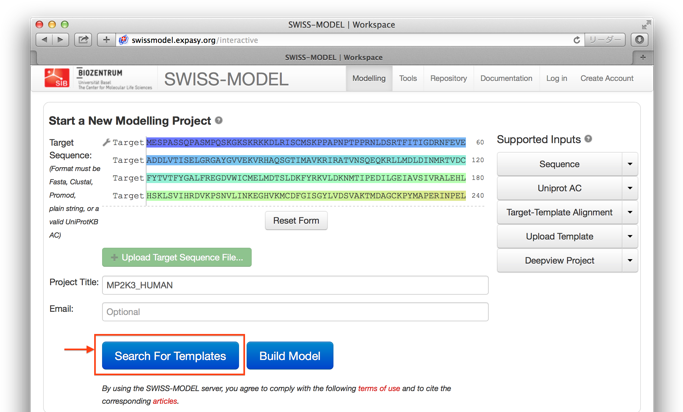
(2)アミノ酸配列が入力されると以下の画面になります。 Project Titleは本解析のプロジェクト名です。ここでは自動的に入力されています。
-
(3)「Search For Templates」をクリックすると、入力したアミノ酸配列からモデルの基になるテンプレートの立体構造データを検索します。
1
次のページでは、モデルをダウンロードする手順をご紹介します。
-
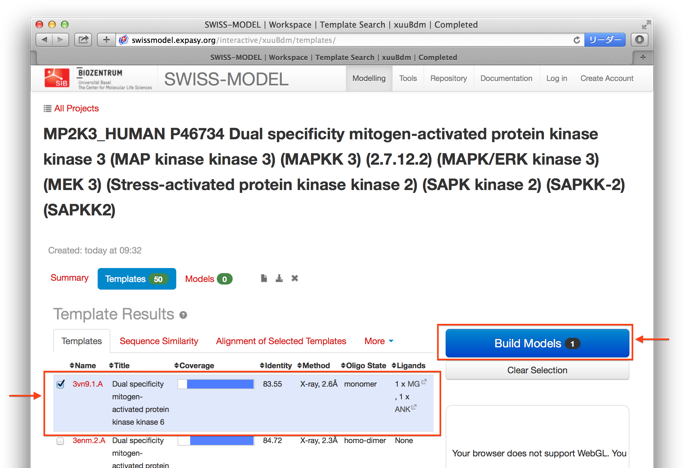
(4)検索が終わるとテンプレート構造の候補が示されます。IdentityやCoverage, Ligands等を参考に、テンプレートに指定したいものを選択します。テンプレート構造を複数指定することも可能です。ここでは、リガンドが結合しているPDB ID:3vn9をテンプレートに選択します。「Build Models」をクリックしてモデリングを開始します。
3. モデリングの結果を見る
-
(1)モデリングが終了すると次の画面になります。Model Resultsには、モデルの品質評価を示すQMEANDisCo Globalのスコア、Ligandの有無、テンプレートとのアミノ酸残基のIdentity、アライメントが表示されます。
-
(2)右側には、構築された立体構造モデルがQMEANの値に基づいて色分け(品質が高いほど青く、赤いほど低く)されて表示されます。
Copyright © 2009-2025 Altif Laboratories Inc. All rights reserved.
