関連情報 SWISS-MODEL の使い方 > SWISS-MODEL Repositoryで構造モデルをダウンロードする
SWISS-MODEL Repositoryは、自動化されたホモロジーモデリングのSWISS-MODELにより生成されたタンパク質立体構造のデータベースです。
詳細は、SWISS-MODEL Repositoryのサイトをご覧下さい。また、お使いになる際には、以下のリファレンスを引用して下さい。
Bienert S, Waterhouse A, de Beer TAP, Tauriello G, Studer G, Bordoli L, Schwede T
The SWISS-MODEL Repository - new features and functionality.
Nucleic Acids Res 45, D313-D319. (2017)
SWISS-MODEL Repositoryで立体構造モデルをダウンロードする
SWISS-MODEL Repositoryについて
赤い線で囲まれた部分に、対象タンパク質のUniProtのAccession CodeやEntry Nameを入力します。
ここでは、ヒトIntestinal-type alkaline phosphataseを
検索するため、Accession Code「P09923」を入力します。
虫眼鏡アイコンをクリックして検索します。
3. 検索結果を見る
SWISS-MODELでは、SWISS-MODELのホモロジーモデリング手法により生成した立体構造モデルを公開しています。
ここでは、そのデータベース、SWISS-MODEL Repositoryからモデルの立体構造をダウンロードする方法を紹介します。
1. SWISS-MODEL Repositoryのページを開く
2. 対象タンパク質のQueryを入力して検索する
入力したタンパク質の立体構造モデルがある場合は、検索結果が表示されます。
SWISS MODELでホモロジーモデリングを行うには
SWISS-MODELのWEBサイトでは、ホモロジーモデリングを行うことができます。モデルを作成したい対象タンパク質のアミノ酸配列とテンプレート構造を入力すると、テンプレート構造に基づき、モデル構造を構築できます。テンプレート構造を指定せずに行うことも可能です。
SWISS-MODELのAutomated Modeでのホモロジーモデリングの手順をこちらのページで紹介しています。
お使いの際には、SWISS-MODELのサイトをご確認ください。
SWISS-MODEL Repository及びAutomated Modeでのモデリングについては、SWISS-MODELより許可を得て、リンク、ご紹介をしております。
SWISS-MODELを使用して変異体のモデル構造を作成し、Waalsを使用して変異による立体構造への影響について解析した例をご紹介しています。ご参照ください。
-
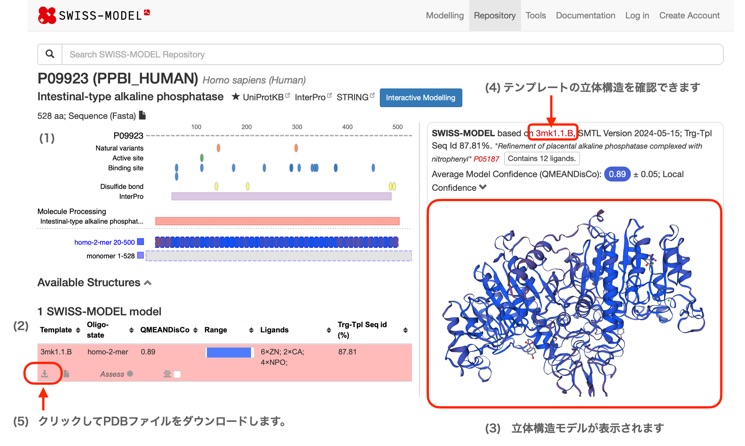
(1) 構造モデルで予測された活性部位や結合部位が示されます。 入力した対象タンパク質のアミノ酸配列の20-500残基に相当します。
-
(2) この構造モデルは、テンプレート構造のPDB ID 3mk1に基づいて生成され、アミノ酸一致度が87.81%、ホモダイマーであること、モデルの信頼度を示すQMEANDisCoは0.89であることが確認できます。リガンドの情報も表示されます。
-
(3) 左側のビューでは、立体構造モデルが表示されます。
-
(4) テンプレートに使用された立体構造を確認できます。
-
(5) ダウンロードアイコンをクリックして立体構造モデルのPDBファイルをダウンロードします。ファイル名はcoords.pdbになります。
Copyright © 2009-2025 Altif Laboratories Inc. All rights reserved.
