関連情報 PDBの使い方 > UniProt経由でPDBファイルをダウンロードする
1. UniProtのエントリーページを開く
UniProt経由でPDBファイルをダウンロードする
UniProtで、タンパク質のエントリーページを開きます。ここでは、ヒトインスリンレセプター(P06213 INSR_HUMAN)のページを示しています。
2. Structureの情報を表示する
UniProtの個々のエントリーのページでは、PDBのデータや、AlphaFoldやSWISS-MODELで予測されたモデル構造のデータを確認できます。
リンクされた各々のデータベースからPBファイルをダウンロードすることができます。
-
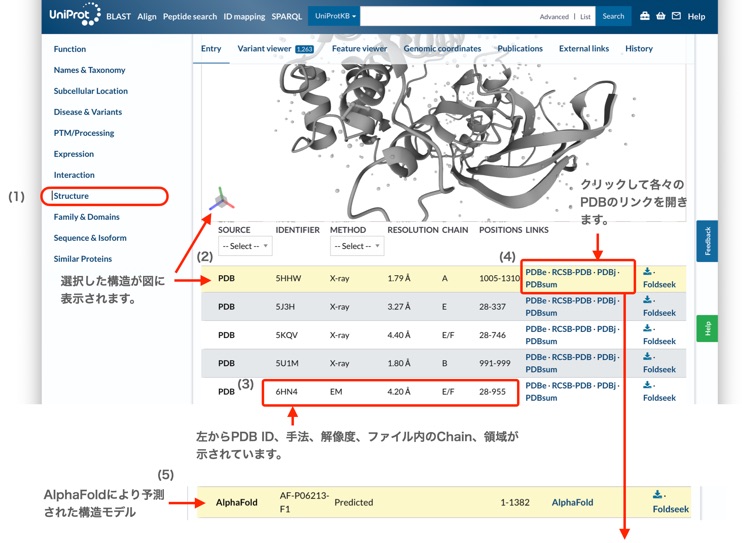
(1)左側に並んだ項目の「Structure」をクリックすると、「Structure」の項目に移動します。
-
(2)PDBデータの一覧が表示されます。選択されたデータの立体構造が上部に表示されます。
-
(3)左から、PDB ID、手法、解像度、ファイル内で相当するChain、アミノ酸配列の領域が示されています。
-
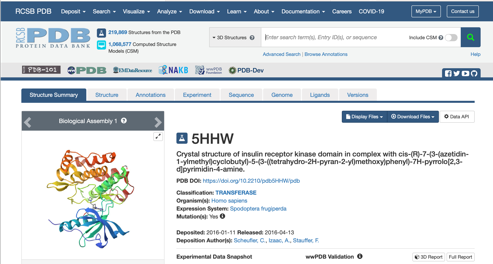
(4)PDBのリンクをクリックしてアクセスします。ここでは、RCSB-PDBの例を示します。
-
(5)AlphaFoldにより既に予測された構造モデルがある場合は、表をスクロールすると確認できます。
2. AlphaFoldやSWISS-MODELで予測された構造モデルをダウンロードする
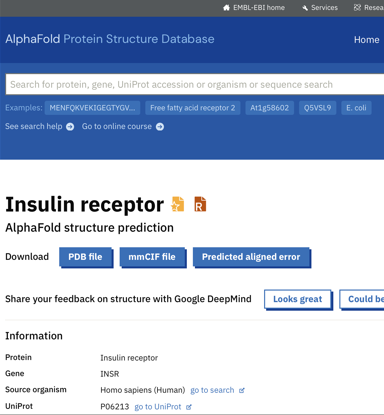
AlphaFold Protein Structure Database (AlphaFoldDB)には、AlphaFoldにより予測された立体構造モデルのデータが登録されています。
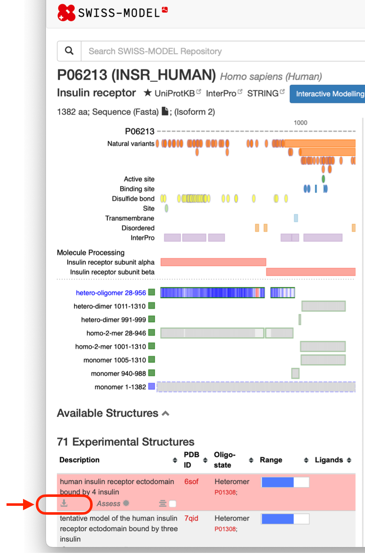
また、SWISS-MODEL Repository (SMR)には、SWISS-MODELのホモロジーモデリングによる立体構造モデルのデータが登録されています。UniProtから、これらの構造データにアクセスできます。
-
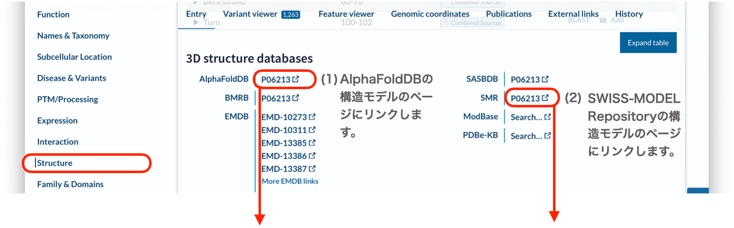
(1)AlphaFoldDBの構造モデルのページが開かれます。PDB Fileをクリックしてダウンロードします。
-
(2)SWISS-MODEL Repositoryの構造モデルのページが開かれます。矢印のアイコンをクリックしてPDBファイルをダウンロードします。
RCSB-PDBのリンク先
AlphaFoldDBからのダウンロードは「 AlphaFold DBから立体構造モデルをダウンロードする」を、SMRからのダウンロードは「 SWISS-MODEL Repositoryでモデル構造をダウンロードする」をご参照ください。
Copyright © 2009-2025 Altif Laboratories Inc. All rights reserved.
