解析事例 > 複数の変異の位置をマップする
1. 立体構造データを入手する
HIV逆転写酵素の立体構造は、Protein Data Bankより入手しました。ここでは、阻害剤が結合していない構造(2HMI)、核酸系阻害剤の一つAZT-triphosphateとの複合体(3V4I)、非核酸系阻害剤の一つnevirapineとの複合体(3V81)を使用しました。
薬剤耐性変異の立体構造へのマッピング HIV逆転写酵素の例
2. 薬剤耐性変異立体構造上の変異の位置
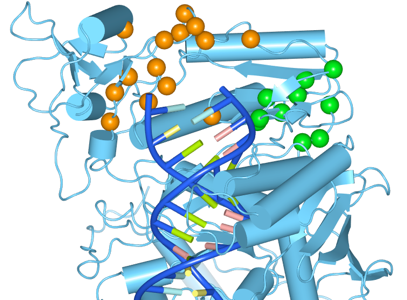
HIV逆転写酵素の立体構造上で、薬剤耐性変異が見られた位置をサイト機能を使ってマップしました。図1は、阻害剤フリーの構造に同定された変異の位置をマップしたものです。核酸系阻害剤に対する耐性変異の位置をOrangeのSpace-fillingで表示しています。非核酸系阻害剤に対する耐性変異はGreenのSpace-fillingで表示しています。核酸系阻害剤、非核酸系阻害剤のそれぞれに対する薬剤耐性変異の位置は、立体構造上局所的に存在することがわかります。
図2は、アミノ酸配列上での変異の位置を示します。 図1と同様、核酸系阻害剤への耐性変異の位置はOrange、非核酸系阻害剤にたいする耐性変異はGreenで示します。アミノ酸配列上では、ばらつきに特に目立った傾向は見られませんが(図2)、立体構造上では、2つのグループにまとまっている様子が見られます。
HIV逆転写酵素の立体構造上に核酸系逆転写酵素阻害剤および非核酸系逆転写酵素阻害剤に対する耐性変異の位置をマップした例をご紹介します。
論文や研究発表では、既知の変異やこれまでの実験結果など、複数のアミノ酸残基を立体構造へマップした図が使われます。アミノ酸残基の位置を立体構造に示すことで、配列では見られなかった立体構造上の位置の傾向が見えてくる場合があります。
核酸系阻害剤に対する耐性変異と非核酸系阻害剤に対する耐性変異の位置を立体構造にマップしたところ、それぞれの変異は構造的に局在していることが確認されました。
図1 HIV逆転写酵素の立体構造上での耐性変異の位置

解析事例

図2 HIV逆転写酵素のアミノ酸配列上での耐性変異の位置
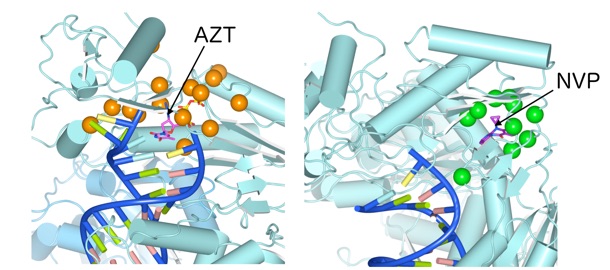
図3 核酸系阻害剤に対する耐性変異(左)と非核酸系阻害剤に対する耐性変異(右)の位置
分子病態解析での使用例 変異による立体構造への影響を調べる
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
PDBデータの向きを揃える Protein Kinase Aの例1
阻害剤との結合様式を比較する Protein Kinase Aの例2
コンフォメーション変化を見る EF-Tuの例
阻害剤との相互作用を調べる PP1と阻害剤Calyculinの例
Bファクターで比較する α-Galactosidaseの例
3. 薬剤耐性変異立体構造上の変異の位置
次に、それぞれのグループを阻害剤との複合体にマップしました。核酸系阻害剤に対する耐性変異は核酸系阻害剤の一つAZTとの複合体に、非核酸系阻害剤に対する耐性変異は核酸系阻害剤の一つNVTとの複合体にマップします。どちらも、耐性変異の位置は、阻害剤の近傍にまとまって存在しているのがわかります。
複数の残基を立体構造にマップすることで、アミノ酸配列上に示したり、表にまとめるだけでは見えてこなかった立体構造上での傾向が見えてくることがあります。既知のデータのまとめやこれまでの実験結果などを立体構造上で考察する際にも有効です。
Copyright © 2009-2025 Altif Laboratories Inc. All rights reserved.