解析事例 > 阻害剤との結合の様式を比較する
1. 1つのPageに2つのPDBデータを読み込む
-
(1)1つのPageに1YDR(左)と1STC(右)のPDBデータを読み込みます(図1)。
-
(2)ここでは、「2つのPDBデータの向きを揃える 」で、予め向きを揃えて前処理したPDBデータを使用しています。
阻害剤との結合の様式を比較する Protein Kinase Aの例2
2. Compound Interfaceで阻害剤近傍の残基を抽出する
-
(1)それぞれのPDBデータでCompound Interfaceを実行します。Cut-off Distanceは4.0[Å]に設定します。
-
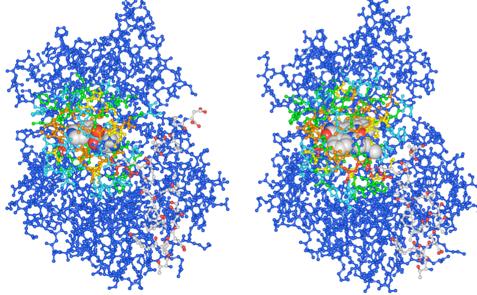
(2)Structure Viewでは、ResiduesのColorがDistanceに変更され、阻害剤からの距離に応じて色分け表示されます(図2)。
3. Workspaceで抽出された構造を見る
-
(1)Workspace Cにはそれぞれ阻害剤から4.0[Å]以内に原子が存在するアミノ酸残基が抽出されています。
6. 全体構造を表示する
-
(1)全体構造を好みの基本表示に指定します。ここでは、Style & Color PanelでResiduesのチェックボックスをOFFにし、CartoonsをONにしてStyleをRibbonsにしています。
-
(2)Main-spaceに切替えます。
-
(3)距離表示を非表示にするには、Label & Distance PanelでDistanceのチェックボックスをOFFにします。
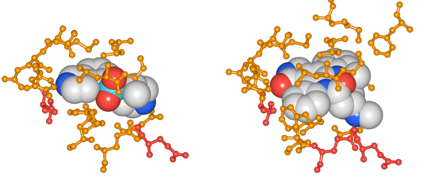
PKAと阻害剤H7(IQP)複合体(左)とPKAとスタウロスポリン (STU) 複合体(右)における阻害剤との水素結合の比較。
阻害剤H7(IQP)とPKAとの間には、Val 123およびGlu 170と2つの水素結合しか見られないが、スタウロスポリン(STU)
との間には、Val 123とGlu 170の他、Glu 121, Glu 127と間に4つの水素結合が見られる。

異なる阻害剤を結合したPDBデータを使用して、阻害剤との結合様式を比較するには、PDBデータの向きを揃えてから、Compound Interfaceを使用します。
ここでは、Protein Kinase A (PKA)の阻害剤H7複合体 (1YDR)とスタウロスポリン複合体 (1STC) を、Compound Interfaceを使用して阻害剤との結合様式を比較する例をご紹介します。
Waals 2013 チュートリアルでは、手順を詳しくご紹介しております。ご参照下さい。
5. 選択して表示
-
(1)インタフェースブラウザから酸素原子と窒素原子の距離の値が3.3[Å] 未満のものを選択し、Workspace Aに取り出します。Stick表示にします。
-
(2)インタフェースブラウザで距離の値が3.3[Å] 未満のものを選択し、DistanceメニューのShow Distance Labelで距離を表示します。ラベルを付けるにはラベルツールを使用します。
4. Interface Browserで抽出結果を見る
-
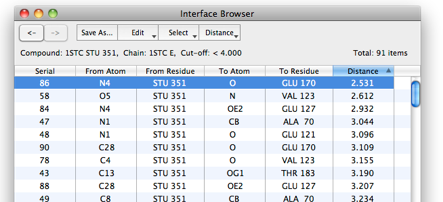
(1)Interface Browserでは、抽出結果を表で確認することができます。
-
(2)Distanceをクリックすると原子間の距離の順で並べ替えられます。抽出結果から、距離の近いものを絞り込むことができます。図4は、1STCの結果を示しています。
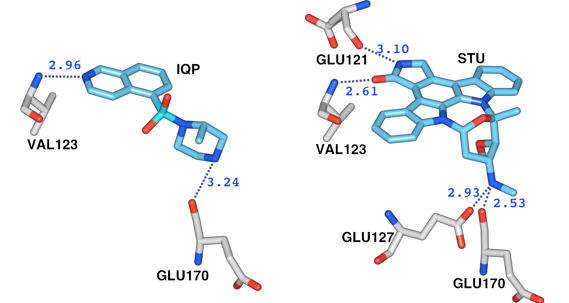
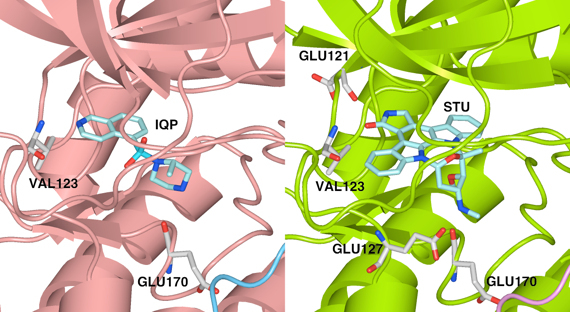
図1
図2
図3
図4
図5
図6 PKAと阻害剤H7(IQP)複合体(左)とPKAとスタウロスポリン (STU) 複合体(右)における阻害剤との水素結合の比較。

解析事例
分子病態解析での使用例 変異による立体構造への影響を調べる
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
PDBデータの向きを揃える Protein Kinase Aの例1
阻害剤との結合様式を比較する Protein Kinase Aの例2
コンフォメーション変化を見る EF-Tuの例
阻害剤との相互作用を調べる PP1と阻害剤Calyculinの例
Bファクターで比較する α-Galactosidaseの例
Copyright © 2009-2013 Altif Laboratories Inc. All rights reserved.