解析事例 > ダイマーインタフェースを抽出する
1. PDBファイルを開く
-
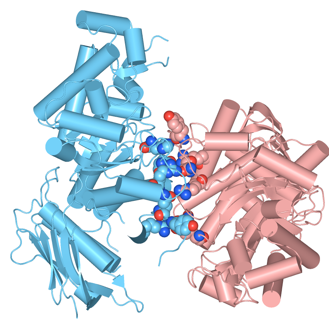
(1)1NOWを開きます。Biological Unit 1のPDBデータを使用しています。サブユニットβのホモダイマー、β-Hexosaminidase B (Hex B)の状態です。
-
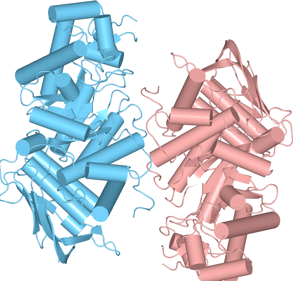
(2)2つのサブユニットの位置関係を把握するために、基本表示をCartoonsのStyleをCylinder & Strand、ColorをChainに、CompoundsをOFFにしています。2つのサブユニットがダイマーを形成しています(図1)。
-
(3)基本表示を変更します。CartoonsをOFFに、ResiduesのStyleを Space-fillingに、ColorをChainにTypeはAll Atomsにします(図2)。2つのサブユニット間のアミノ酸残基が相互作用しているのが確認できます。
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
2. Chain Interfaceでサブユニット間の残基を抽出する
-
(1)メニューのTools > Chain Interface...を選択し、Chain Interfaceダイアログを開きます。
-
(2)Cut-off Distanceは3.0[Å]に設定します。
-
(3)Findをクリックします。
-
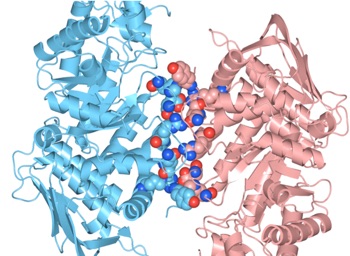
(4)Structure Viewでは、ResiduesのColorがDistanceに変更され、互いにサブユニットからの距離に応じて色分け表示されます(図3)。
3. Workspaceで抽出された構造を見る
-
(1)Workspace Cにはサブユニット間で3.0[Å]以内に原子が存在するアミノ酸残基が抽出されています。
-
(2)ここでは、Contents Ind View内のStyle & Color Panelで、一方のサブユニットでResiduesのColorをAtom Color Carbon With > light blue、もう一方のサブユニットをAtom Color Carbon With >light redに設定しています。
-
(3)ラベルツールでラベルを付けます(図8)。
Chain Interface機能を使用して、ダイマーインタフェースを検出することができます。
目で確認して探すのには難しい、サブユニット間に存在するアミノ酸残基を網羅的に見つけることができます。
ここでは、ホモダイマーのβ-Hexosaminidase B (1NOW)を使用し、2つのサブユニット間で3.0 [Å] 以内の原子を含むアミノ酸残基を抽出する例をご紹介します。
6. 距離を表示
-
(1)Stick表示にします。Workspace内の残基を⌘+Aで全てを選択し、メニューのStructure > Residues > Style > Stickを選びます。
-
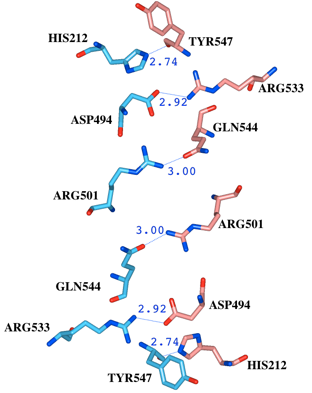
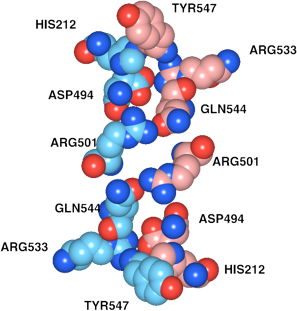
(2)インタフェースブラウザで、抽出された6つの原子のペアを全て選択し、DistanceメニューからShow Distance Labelを選択して、Structure Viewに距離を表示します(図9)。
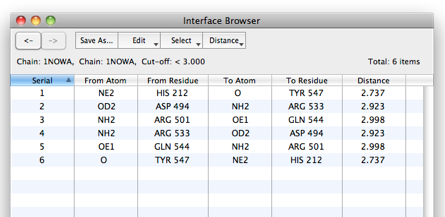
4. Interface Browserで抽出結果を見る
-
(1)Interface Browserでは、抽出結果を表で確認することができます(図7)。
-
(2)サブユニット間で3.0[Å]以内に存在する原子のペアが6つ抽出されました。抽出された残基や原子、原子間の距離が確認できます。
図1
図2
図3
図5
図6
図8
図9 Hexosaminidase Bの2つのサブユニット間で3.0[Å]以内に存在しているアミノ酸残基とその結合の様子。

3. モノマーの構造で接触面を見る
-
(1)ダイマーのうちの右側のChainを非表示にします。Contents Into View内のStyle & Color PanelでChain A No.1のResiduesのチェックボックスをOFFにします(図4)。
-
(2)接触面を見るために90度回転します。メニューのView > Rotate...でRotateダイアログを開きます。
-
(3)Y-axisに-90を入力し、Applyをクリックします。構造がY軸方向に-90度回転し、接触面を見る事ができます(図5)。
-
(4)色が赤いほどもう一方のサブユニットに近いことを示します。赤で示された残基は相手のサブユニットから3.0[Å]以内に原子が存在するアミノ酸残基です。
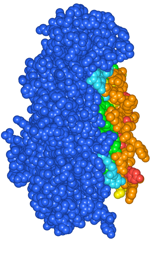
図4
4. 抽出結果をアミノ酸配列で確認する
抽出されたアミノ酸残基距離による色分け表示は、アミノ酸配列の色に反映しています(図6)。立体構造上で一つの面にまとまって存在している領域が、配列上では、いくつかの領域に分かれて存在する様子がわかります。相手のサブユニットから3.0[Å]以内の距離に存在している残基は赤で示されています。
Chain Interfaceの使い方は、Waals ユーザーズガイド
「第8章 解析機能を使う」内の「Chain InterfaceでChain間のアミノ酸残基を抽出する」(107ページ)をご参照ください。
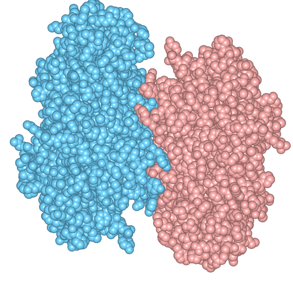
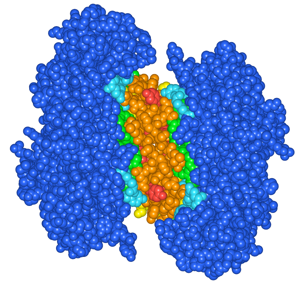

図7

解析事例
分子病態解析での使用例 変異による立体構造への影響を調べる
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
PDBデータの向きを揃える Protein Kinase Aの例1
阻害剤との結合様式を比較する Protein Kinase Aの例2
コンフォメーション変化を見る EF-Tuの例
阻害剤との相互作用を調べる PP1と阻害剤Calyculinの例
Bファクターで比較する α-Galactosidaseの例
Copyright © 2009-2013 Altif Laboratories Inc. All rights reserved.