解析事例 > 阻害剤との相互作用を調べる
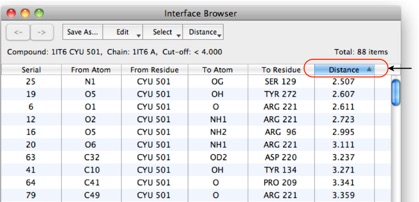
5. 抽出結果を表で確認する
Interface Browserには、CYUの各原子から指定された4.0[Å]以内に原子が存在するアミノ酸残基と原子間の距離が表示されます。
Distanceをクリックして距離で並べ替えます。原子の種類や二原子間の距離を参考にして、CYUの結合に関与しているアミノ酸残基を絞り込むことができます。
阻害剤との相互作用を調べる Protein Phosphatase 1 の例
1. PDBファイルを開く
-
(1)プロテインホスファターゼPP1と阻害剤カリキュリン(CYU)の複合体1IT6のPDBファイルを開きます。ここでは、Chain Aのみ表示します。
-
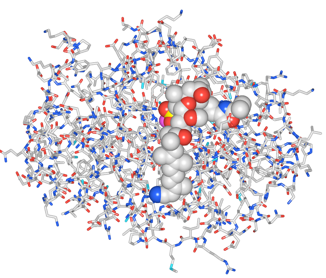
(2)Style & Color Panelで、基本表示を設定します。ここでは、PP1の全原子を表示するため、ResiduesのStyleをStickに、TypeはAll Atomsにします。CompoundsのStyleをSpace-fillingにします(図1)。
Compound Interfaceを使用して、リガンド近傍のアミノ酸残基を抽出することができます。指定した、結合物の全ての原子から指定した距離範囲に存在する原子を含むアミノ酸残基が全て抽出されます。
ここでは、プロテインホスファターゼ1(PP1)と阻害剤カリキュリン(CYU)の複合体であるPDBデータを使用して、CYUの全て原子から4.0[Å]以内の原子を含むアミノ酸残基を抽出し、CYUと相互作用するアミノ酸残基を調べます。
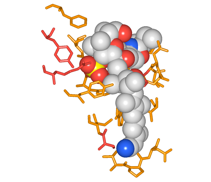
3. 抽出結果を構造で確認する
-
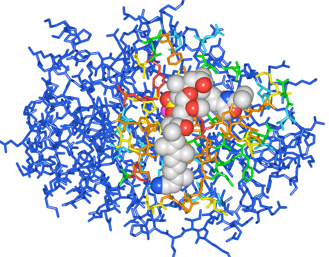
(1)抽出されたアミノ酸残基の構造を表示するには、Structure Viewの表示をWorkspace Cに切り替えます。
-
(2)3.0-4.0[Å]の残基はオレンジで、3.0[Å]未満の残基は赤で示されます。
5. 解析結果の図を他の書類に貼り込む
Structure Viewに表示された立体構造のイメージをコピーするには、メニューのEdit > Copyを選択するか、Structure Viewにマウスを入れた状態で⌘+Cキーを押します。Contents Info Viewにマウスを入れた状態で ⌘+Cキーを押すとアミノ酸配列の表示をPDFでコピーできます。 他の書類でペーストします。


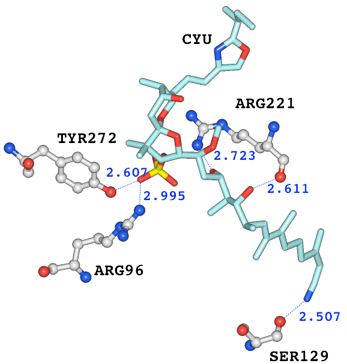
6. インタフェースブラウザから選択する
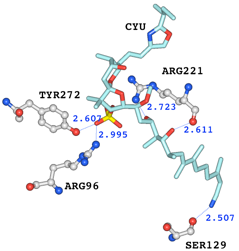
インタフェースブラウザから酸素原子と窒素原子の距離の値が3.0[Å] 未満のものを選択し、Workspace Aに取り出しました。インタフェースブラウザからは、選択した結果の距離表示をすることができます。ラベルを付けるにはラベルツールを使用します。
4. 抽出結果をアミノ酸配列で確認する
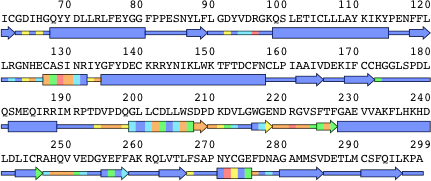
抽出されたアミノ酸残基距離による色分け表示は、アミノ酸配列の色に反映しています。配列上では、R96, S129, R221, Y272が赤色で示され、阻害剤から3.0[Å]未満の距離に存在していることがわかります。
図5 Contents Info Viewのアミノ酸配列。ChainsのColorをDistanceにしています。
2. Compound Interfaceを実行する
-
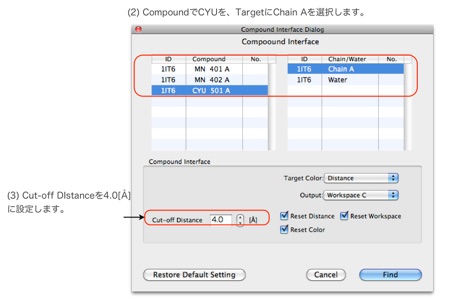
(1)メニューのTools > Compound Interface...を選択し、Compound Interfaceダイアログを開きます(図2)。
-
(2)CompoundでCYUを、TargetにChain Aを選択します。
-
(3)Cut-off Distanceで、距離を4.0[Å]に指定します。
-
(4)Findをクリックします。
-
(5)Structure Viewの表示が距離による色分け表示に切り替わります(図3)。CYUからの距離が近い程赤く、遠いほど青く表示されます。
図1
図2 Compound Interfaceダイアログ
図3 阻害剤CYUから距離による色分け表示
図4 抽出された阻害剤近傍のアミノ酸残基
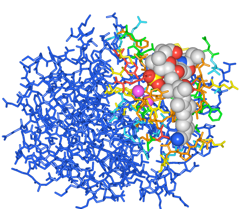
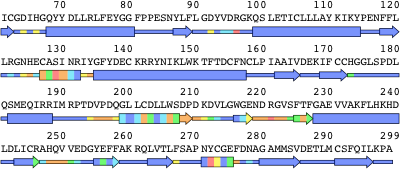
図6 抽出されたアミノ酸残基と阻害剤の原子間距離

解析事例
分子病態解析での使用例 変異による立体構造への影響を調べる
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
PDBデータの向きを揃える Protein Kinase Aの例1
阻害剤との結合様式を比較する Protein Kinase Aの例2
コンフォメーション変化を見る EF-Tuの例
阻害剤との相互作用を調べる PP1と阻害剤Calyculinの例
Bファクターで比較する α-Galactosidaseの例
Copyright © 2009-2013 Altif Laboratories Inc. All rights reserved.