解析事例 > 金属イオンと結合する残基を検出する
金属イオンと結合する残基を検出する Protein Phosphatase 2Cの例
1つの原子と結合するアミノ酸残基を調べるには、球ピックによる選択が便利です。選択した原子から空間的に距離で選択することができます。
PYL proteinとプロテインホスファターゼ2C (PP2C)複合体(3KDJ)のPP2CにはMnイオンが結合しています。ここでは、球ピックを使用して、Mnイオンから3.0[Å]以内の原子を含むアミノ酸残基を選択し、調べる手順をご紹介します。
1. PDBファイルを開く
-
(1)3KDJ を開きます。ここでは、PP2Cに相当するChain Bのみ表示しています。
-
(2)Style & Color Panelで、基本表示を設定します。ここでは、ResiduesのStyleをBall & Stickに、TypeはAll Atomsにします。CompoundsのStyleをSpace-fillingにします(図1)。
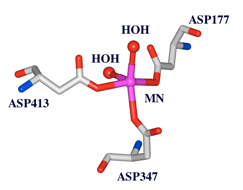
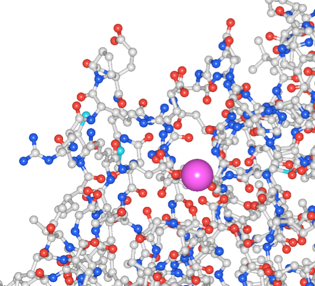
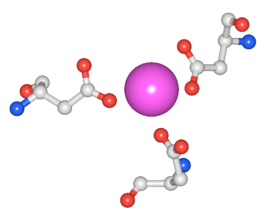
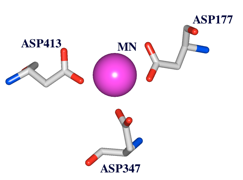
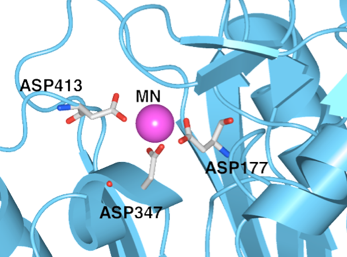
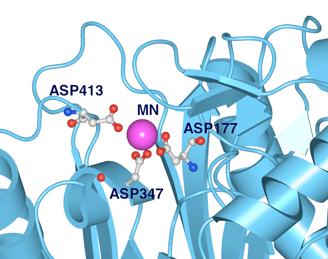
図2 Workspace Aに取り出した状態
図3 ラベルを貼り、ResiduesをStickで表示
図4 水分子の酸素を表示。CompoundsをStickで表示すると、PDBに結合状態が記載されている場合はボンドが表示されます。
2. 球ピックツールで選択する
-
(1)Structure Viewのツールアイコンから球ピックツールを選択します。マウスでMnをクリックし、ドラッグして3.0[Å]以内のアミノ酸残基を選択します。
-
(2)3つのアミノ酸残基の構造が選択されます。
3. Workspaceに取り出す
-
(1)メニューのWorkspace > Add into Workspace Aを選択し、Workspace Aに取り出します。
-
(2)Workspace Aに切替えると、取り出した構造だけを見る事ができます(図2)。
図1 PP2Cに結合しているMn (Magenta)
4. Workspaceで詳細を見る
-
(1)Workspace Aに取り出した構造は、Mnイオンと3残基のアミノ酸なので、回転させて互いの位置関係を見たり、ラベルを付ける操作も簡単です(図3)。
-
(2)球ピックでは、水分子も選択されます。水分子を表示するには、Style & Color PanelでWaterのチェックボックスをONにします(図4)。水分子がMnに配位しているのがわかります。
5. 表示形式を設定する
-
(1)Mnと結合している3つのアミノ酸残基を指定表示に設定します。3つの残基を選択し、メニューのStructure > Residues > Style > Stickを選択します。
-
(2)全体構造を基本の表示形式で表示します。ここでは、Style & Color PanelでResiduesのチェックボックスをOFFにし、ChainsをONにしてStyleをRibbons、ColorをLight Blueにしています。
-
(3)Main-spaceに切替えて全体を表示します(図5)。
図5 全体をRibbonsで表示し、検出したアミノ酸残基をStickで表示

解析事例
分子病態解析での使用例 変異による立体構造への影響を調べる
ダイマーインタフェースを抽出する β-Hexosaminidase Bの例
PDBデータの向きを揃える Protein Kinase Aの例1
阻害剤との結合様式を比較する Protein Kinase Aの例2
コンフォメーション変化を見る EF-Tuの例
阻害剤との相互作用を調べる PP1と阻害剤Calyculinの例
Bファクターで比較する α-Galactosidaseの例
Copyright © 2009-2013 Altif Laboratories Inc. All rights reserved.